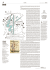strumenti di analisi multivariata per lo studio e la classificazione di
Transcript
strumenti di analisi multivariata per lo studio e la classificazione di
“STRUMENTI DI ANALISI MULTIVARIATA PER LO STUDIO E LA CLASSIFICAZIONE DI MAPPE 2D-PAGE PER L’ANALISI DEL PROTEOMA” E. Marengo*, E. Robotti*, P.G. Righetti** * Dipartimento di Scienze dell’Ambiente e della Vita - University of Eastern Piedmont Spalto Marengo 33 - 15100 Alessandria - Italy - e-mail: [email protected] ** Scientific and Technological Department - University of Verona - Strada le Grazie 15 Verona - Italy La gel-elettroforesi bidimensionale è una delle tecniche più diffuse nei campi della genomica e proteomica, per lo studio delle proteine presenti in estratti cellulari umani, animali o vegetali. Lungo la prima dimensione, le proteine sono separate in base al punto isoelettrico (attraverso un gradiente di pH), mentre la seconda dimensione ne esegue una successiva sulla base della massa molecolare (attraverso un gradiente di porosità). Il risultato finale è una mappa bidimensionale, chiamata 2D-PAGE (2D polyacrilamide gel-electrophoresis), sulla quale le proteine sono rappresentate come macchie disperse nella matrice polimerica. Il patrimonio proteico cellulare può andare incontro a profonde modificazioni (cambiamenti delle abbondanze relative di alcune proteine; presenza/assenza di altre proteine) in seguito all’insorgere di una patologia oppure in seguito alla somministrazione di un farmaco e questo ne giustifica l’interesse e l’importanza. Non a caso, il confronto di mappe 2D-PAGE appartenenti a classi diverse di campioni (sani/malati oppure malati/trattati con un farmaco) appare fondamentale al fine di utilizzare le stesse mappe 2D per scopi diagnostici/prognostici oppure nell’ambito di studi di drug-design; questo utilizzo è purtroppo reso problematico dalla scarsa riproducibilità che affligge la tecnica analitica. La bassa ripetibilità è dovuta ad alcune caratteristiche quali: 1) la complessità del campione (biologico); 2) la complessità dei passaggi di pre-trattamento del campione; 3) il numero elevato di parametri strumentali che devono essere controllati durante la corsa elettroforetica. La variabilità è tanto elevata da causare differenze anche molto marcate all’interno di mappe replicate della stessa corsa elettroforetica. Alla luce di queste considerazioni, appare fondamentale la messa a punto di nuovi metodi, che permettano di tenere in considerazione la scarsa riproducibilità e che consentano di eseguire confronti più efficaci tra le diverse classi di mappe 2D-PAGE. Il nostro gruppo di ricerca ha sviluppato metodi per il confronto di mappe 2D-PAGE, attraverso l’utilizzo di diversi strumenti della statistica multivariata. Sono stati applicati con successo metodi basati sull’Analisi delle Componenti Principali, accoppiata con metodi di classificazione (Linear Discriminant Analysis e SIMCA), sulla Three-Way PCA, sul Multidimensional Scaling e sui principi della logica fuzzy. Tutti questi approcci permettono di tenere in considerazione la scarsa riproducibilità della tecnica sperimentale e sono stati applicati con successo a svariati dataset. Di recente, un approccio basato sull’Analisi delle Componenti Principali e sul metodo SIMCA (Soft-Independent Model of Class Analogy) è stato applicato con successo a due dataset di natura diversa. Il primo è costituito da mappe 2D-PAGE relative a estratti proteici di ghiandole surrenali di topolini sani e affetti da neuroblastoma. Il neuroblastoma, insieme a linfoma, leucemia, osteosarcoma, tumori di Ewing, rabdomiosarcoma e leucemia linfoblastica, appartiene a un gruppo di tumori infantili non differenziati conosciuti come tumori a cellule piccole. Il neuroblastoma rappresenta la più comune forma tumorale extracranica solida dell’infanzia; colpisce il midollo surrenale o i gangli del sistema simpatico di addome, petto o collo. Questa gravissima patologia è responsabile di circa il 96% dei tumori infantili. L’analisi delle componenti principali è stata applicata a 20 mappe (a) (b) 2D-PAGE relative a estratti proteici di ghiandole surrenali di topolini sani (10 mappe) e affetti da neuroblastoma (10 mappe). In figura 1 è rappresentato un esempio di mappa 2D-PAGE ottenuta per ciascuna classe di campioni. Lo score plot relativo alle prime due componenti principali è riporato in figura 2 e mostra i due gruppi di campioni separati efficacemente lungo la prima componente Figura 1: Esempi di mappe 2D-PAGE: campione di controllo (a) e campione affetto da Neuroblastoma (b) principale. Successivamente è stato applicato il metodo SIMCA per identificare le classi di campioni presenti e le macchie responsabili delle differenze riscontrate. Tra le catene polipeptidiche che sono risultate sovra- o sotto- espresse, alcune sono state identificate attraverso la spettrometria di massa MALDI-TOF. Figura 2: Score plot PC2 vs PC1 (campioni di controllo (HEA1-HEA10); campioni patologici (ILL1ILL10) ) Il secondo dataset è invece relativo alla messa a punto di nuove linee cellulari utilizzate come standard di riferimento. Nell’ottica dello sviluppo di una nuova linea cellulare standard, si rende necessario il confronto tra il pattern bidimensionale fornito dalla linea cellulare già in commercio con quello generato dalla linea cellulare che si vuole mettere a punto. Il confronto è stato eseguito su 10 mappe 2D-PAGE relative alle due linee. L’analisi delle componenti principali e la successiva applicazione del metodo SIMCA hanno permesso di valutare le differenze esistenti tra i due tipi di pattern bidimensionale. Attualmente si sta procedendo all’identificazione delle catene polipeptidiche responsabili delle differenze riscontrate.